Stan Advent Boot Camp 第9日目: 階層線形モデルをやってみよう
この記事について
Stan Advent Calendar 2020 9日目の記事です。本記事で紹介する階層線形モデルは、第4日目に紹介された重回帰モデルや、第5日目に紹介されたロジスティック回帰モデルなどの、一般線形モデルや一般化線形モデルを拡張したモデルになります。Stanで階層線形モデルの確率モデルを書く方法を紹介します。
なおこの記事では階層線形モデルという名称を使用しますが、線形混合モデルやマルチレベルモデルとも本質的には同じです。
一般線形モデルの復習
ここでは説明変数が1つの、単回帰モデルを例とします。
個のデータがあり、目的変数
を、説明変数
で予測しています。
が切片、
が回帰係数、
が残差です。
これは以下の確率モデルと同義です。
つまり、
というモデルを立てたことになります。
階層線形モデルへの拡張
データの階層性を考慮すべき状況
StanとRでベイズ統計モデリングの121ページから引用すると、階層モデルとは
説明変数だけでは説明がつかない、グループに由来する差異(グループ差)を上手く扱うための一手法
です。
「グループ」というのは例えば、
| 実験参加者ID | グループ |
|---|---|
| 1 | ◇◇大学 |
| 2 | ◇◇大学 |
| 3 | ◇◇大学 |
| 4 | ▲▲大学 |
| 5 | ▲▲大学 |
| 6 | ▲▲大学 |
| 7 | ☆☆大学 |
| 8 | ☆☆大学 |
| 9 | ☆☆大学 |
などです。グループは上表のような「所属集団」に限らず、以下のように個人をグループと見なすことも可能です。下表では、「実験参加者ID」がグループに相当しています。
| 実験参加者ID | 試行番号 |
|---|---|
| 1 | 1 |
| 1 | 2 |
| 1 | 3 |
| 2 | 1 |
| 2 | 2 |
| 2 | 3 |
| 3 | 1 |
| 3 | 2 |
| 3 | 3 |
上の2つの表ではいずれも、9つのデータが得られていることになっています。では、目的変数と説明変数
も同様に9つずつデータが得られているとき、
という回帰モデルを考えてよいかというと...そうとは限りません。ここで、データの階層性を考慮しなければならない場合があります。その理由は、以下のスライドの12枚目に詳しいです。
階層線形モデルの確率モデル
上記の回帰モデルを階層線形モデルに拡張したい場合、色々な拡張の方法がありますが、典型的には以下の3通りが考えられます。
- 切片のみに、グループ差を考慮する
- 回帰係数のみに、グループ差を考慮する
- 切片と回帰係数に、グループ差を考慮する
ここでは一例として、「3. 切片と回帰係数に、グループ差を考慮する」場合の階層線形モデルの確率モデルを紹介します。
添え字は一つひとつのデータを、添え字
は一つひとつのグループを表します。上の表でいえば(一つ目の表)、添え字
は一人一人の実験参加者を、添え字
は各大学を表しています。切片パラメータ
と回帰係数パラメータ
に添え字
が付いて、
と
になっているので、グループごとにこれらのパラメータが異なることを仮定しています。
では、グループごとに異なると仮定した切片と回帰係数は、どのように、どれくらいグループごとに異なるのでしょうか。それを反映しているのが、
です。つまりグループごとの切片と回帰係数の違いは、正規分布に従うというモデルを立てたことになります。
実演
まずは、パラメータの真値と真のモデルが分かっている状況からサンプルデータを生成します。
library(ggplot2) library(rstan) set.seed(1234) n_data = 100 #総データ数 n_group = 5 #グループ数 n_each_group = n_data / n_group #各グループ内の人数 = 20 mu_0 = rnorm(n = n_group, mean = 30, sd = 10) #切片は、平均30、標準偏差10の正規分布に従う mu_1 = rnorm(n = n_group, mean = 3, sd = 1.5) #回帰係数、平均3、標準偏差1.5の正規分布に従う x = runif(n = n_data, min = 1, max = 30) #説明変数 y = rnorm(n = n_data, mean = (mu_0 + mu_1*x), sd = 15) #目的変数 group = rep(1:n_group, time = n_each_group) #グループを識別する変数
このようなサンプルデータの作り方は、StanとRでベイズ統計モデリングに載っています。
グループごとに回帰直線を描くと、こんな感じです。確かに切片も傾きも、グループごとに異なっています。
ggplot(data = data.frame(x, y, group), mapping = aes(x = x, y = y, color = factor(group))) + geom_point() + geom_smooth(method = "lm", se = FALSE)

この確率モデルを指定してパラメータを推定したら、真値に近くなることが期待できるはずです。やってみましょう。Stanコードは以下です。
方法1:回帰モデルを拡張した書き方
data {
int<lower=0> n_data; //データ数
int<lower=0> n_group; //グループ数
real y[n_data]; //目的変数
vector[n_data] x; //説明変数
int group[n_data]; //グループを識別する変数
}
parameters {
vector[n_group] beta[2]; //beta[1]が各グループの切片
//beta[2]が各グループの回帰係数
real mu[2]; //mu[1]が各グループの切片の平均
//mu[2]が各グループの回帰係数の平均
real<lower=0> sigma[3]; //sigma[1]が各グループの切片の標準偏差
//sigma[2]が各グループの回帰係数の標準偏差
//sigma[3]が目的変数が従う正規分布の標準偏差
}
model {
beta[1] ~ normal(mu[1], sigma[1]);
beta[2] ~ normal(mu[2], sigma[2]);
y ~ normal(beta[1, group] + beta[2, group].*x, sigma[3]);
}
.*という演算子が用いられていることに注意してください。これは同じ長さのベクトルを、要素ごとに掛算するための演算子です。
これをmixed_1.stanという名前で保存して、以下のRコードからサンプリングを行います。
sm_1 = rstan::stan_model("mixed_1.stan") fit_1 = rstan::sampling(object = sm_1, data = list( n_data = n_data, n_group = n_group, y = y, x = x, group = group ), seed = 1234, iter = 4000, warmup = 1000) print(fit_1)
トレースプロットやchainごとの事後分布を視覚的にチェックした限りでは、収束していると判断して良さそうです。

事後分布の要約統計量は以下の通りです。

- グループごとの切片が従う正規分布の平均
]は、真値が30のところ、事後中央値は28.27
- グループごとの切片が従う正規分布の標準偏差
]は、真値が10のところ、事後中央値は18.26
- グループごとの回帰係数が従う正規分布の平均
]は、真値が3のところ、事後中央値は2.86
- グループごとの回帰係数が従う正規分布の標準偏差
]は、真値が1.5のところ、事後中央値は1.62
- 目的変数が従う正規分布の標準偏差
]は、真値が15のところ、事後中央値は15.22
グループごとの切片が従う正規分布の標準偏差]だけちょっと真値と事後中央値が離れていますが、おおむね良くパラメータリカバリできているように思います。
方法2:多変量正規分布を用いた書き方
上記の階層モデルは、多変量正規分布を用いて、以下のように書くこともできます。多変量正規分布は、Stan Advent Calendar 2020 第6日目の記事でも紹介されています。
※前述の確率モデルに対して、違うStanコードの書き方ができるということではなく、確率モデルも多変量正規分布を用いた別のものに変わっていることに注意してください
data {
int<lower=0> n_data; //データ数
int<lower=0> n_group; //グループ数
int<lower=0> n_slope; //グループ差を考慮する回帰係数の数(切片含む)
real y[n_data]; //目的変数
matrix[n_data, n_slope] X; //説明変数(切片含む)の行列
int group[n_data]; //グループを識別する変数
}
parameters {
vector[n_slope] beta[n_group]; //グループごとの、回帰係数(切片含む)のベクトル
real<lower=0> sigma; //目的変数が従う正規分布の標準偏差
vector[n_slope] mu; //各回帰係数(切片含む)の平均ベクトル
cov_matrix[n_slope] Cov; //多変量正規分布の分散共分散行列
}
model {
for(n in 1:n_data){
y[n] ~ normal(X[n]*beta[group[n]], sigma);
}
beta ~ multi_normal(mu, Cov);
}
modelブロックのy[n] ~ normal(X[n]*beta[group[n]], sigma)は、行列の掛算によって、目的変数が従う正規分布の平均を計算しています。重回帰モデルにおける同様の書き方は、Stan Advent Calendar2020の4日目の記事でも紹介されています。
ポイントは、各回帰係数(切片を含む)がvector[n_slope] beta[n_group]で宣言されているところです。回帰係数の数(切片を含む)の要素を持つbetaというパラメータが、グループの数(n_group)だけあると宣言しています。
modelブロックのbeta ~ multi_normal(mu, Cov)は、各回帰係数(切片を含む)betaが、多変量正規分布に従うことを仮定しています。多変量正規分布のパラメータは、以下の2つです。
- 平均ベクトル
mu - 分散共分散行列
Cov
要するに、それぞれの回帰係数は同時に正規分布に従っており、かつ互いに相関があるというモデルになっています。
ただし多変量正規分布の分散共分散行列を、quad_form_diag()というStanの関数を用いて以下のように定義すると、サンプリング効率がよくなるようです。
data {
int<lower=0> n_data; //データ数
int<lower=0> n_group; //グループ数
int<lower=0> n_slope; //グループ差を考慮する回帰係数の数(切片含む)
real y[n_data]; //目的変数
matrix[n_data, n_slope] X; //説明変数(切片含む)の行列
int group[n_data]; //グループを識別する変数
}
parameters {
vector[n_slope] beta[n_group]; //グループごとの、回帰係数(切片含む)のベクトル
real<lower=0> sigma; //目的変数が従う正規分布の標準偏差
vector[n_slope] mu; //各回帰係数(切片含む)の平均ベクトル
corr_matrix[n_slope] Omega; //それぞれの正規分布同士の相関行列
vector<lower=0>[n_slope] tau; //それぞれの正規分布の標準偏差
}
model {
for(n in 1:n_data){
y[n] ~ normal(X[n]*beta[group[n]], sigma);
}
beta ~ multi_normal(mu, quad_form_diag(Omega, tau));
Omega ~ lkj_corr(2); //相関行列の弱情報事前分布。
}
Stan モデリング言語: ユーザーガイド・リファレンスマニュアル(日本語翻訳版)によればquad_form_diag()という関数は、
quad_form_diag(Sigma,tau)がdiag_matrix(tau) * Sigma * diag_matrix(tau)と等価になるように定義されています. ここで,diag_matrix_(tau)は, 対角成分がtauとなり, それ以外が0の行列を返します
という働きを持ちます(上記のStanコードでは、SigmaというパラメータをOmegaという名前で宣言しています)。
これで本当に分散共分散行列が作れることを確かめてみましょう。今回のように、グループ差を仮定している回帰係数が2つの場合(切片と、説明変数の回帰係数の2つ)、以下のような計算になります。

対角成分は、標準偏差に相当するの2乗なので、分散を表します。非対角成分は、標準偏差
の積に、相関係数
を掛けているので、共分散を表します。確かに、分散共分散行列が作れていますね。
それでは上記のStanコードをmixed_2.stanという名前で保存し、以下のRコードを実行してみましょう。使用しているサンプルデータは、mixed_1.stanに渡したものと同じです。
model.matrix()は、引数にformula(回帰式)を指定すると、説明変数を行列形式でまとめてくれる関数です。今回は切片も回帰係数とみなしているので、全ての値が1の説明変数を追加する必要があるため、X = model.matrix(y ~ 1 + x)という書き方にしています(ただしX = model.matrix(y ~ x)でも同じです)。
X = model.matrix(y ~ 1 + x) # = の左側は大文字のX、右側は小文字のxであることに注意 fit_2 = rstan::sampling(object = sm_2, data = list( n_data = n_data, n_group = n_group, n_slope = ncol(X), y = y, X = X, # = の左右ともに、大文字のXであることに注意 group = group ), seed = 1234, iter = 4000, warmup = 1000)
パラメータ数が多いので、トレースプロットやチェインごとの事後分布は掲載しませんが、収束していると判断して良さそうです。事後分布の要約統計量は以下の通りです。

- グループごとの切片が従う正規分布の平均
]は、真値が30のところ、事後中央値は28.15
- グループごとの切片が従う正規分布の標準偏差
]は、真値が10のところ、事後中央値は19.41
- グループごとの回帰係数が従う正規分布の平均
]は、真値が3のところ、事後中央値は2.85
- グループごとの回帰係数が従う正規分布の標準偏差
]は、真値が1.5のところ、事後中央値は1.67
- 目的変数が従う正規分布の標準偏差
は、真値が15のところ、事後中央値は15.20
相変わらず、グループごとの切片が従う正規分布の標準偏差]だけちょっと真値と事後中央値が離れていますが、おおむね良くパラメータリカバリできていますね。
まとめ
いかがだったでしょうか
パラメータに階層性を仮定したモデルは、ちょっとだけ書き方が難しくなりますが、基本的には回帰モデルの拡張と考えられます。実際のデータ分析では、階層性を考慮したほうがよい状況も多いので、参考になれば幸いです。
Enjoy!
昔はHarukara.Rというのがあったんだが、膝に矢を受けてしまってな
この記事について
ベイズ塾 Advent Calendar 2020 5日目の記事です。このブログは技術ブログ・書評が中心なんですが、今回の記事はポエムです。
広島ベイズ塾は、創設メンバーの一人、某氏(ここでは仮にA氏とする)が広島にいらっしゃったときにできました。詳しい話は、もしかしたら最終日(2020/12/25)に紹介されるのかもしれません。
磯野ー、カラオケしようぜー
しばらくして、A氏が関西に異動されました。カラオケに誘われました。
A氏「カラオケの前に寿司でも食うか」
僕「おっ、いいですねー。あ、でもどこも混んでますねー。ググったら、近くに1件だけ寿司屋ありました、もうそこにしますか」
A氏「せやな」
繁華街の片隅にひっそりと佇むその店内には、ショーケースに色んなネタの切り身が陳列されていた。ネタの名前も、価格も表示されていない。名もなき詩魚(うお)。
僕「(ひそひそ、Aさん、これ、何のネタやと思います?)」
A氏「(ひそひそ、わからへん。赤いな)」
ええい、残さずに全部食べてやる oh darlin 君は誰
そんなこんなで僕らの間には、「寿司を食べてからカラオケに行く」という謎のルーティーンが出来あがったのでした。僕らがよく行っていた寿司屋の名前(はる〇〇)と、カラオケを合わせて、Harukaraの誕生です。二人組音楽ユニットじゃありません(いやそれHALCALI)
Rの勉強会でもする?
時を同じくして広島では、R旋風が巻き起こっていました。その中心はHijiyama.RというRの勉強会(僕らも準レギュラーでしたけど)。
関西でもやる? どちらからともなくそう切り出したのは、偶然ではなく必然だったのかもしれない。
そんなわけで、Rの勉強会をしてから寿司を食べてカラオケに行く、Harukara.Rが誕生しました。
まあRの勉強会といいつつ、その実態は統計の勉強会でした。Rを使って計算をしているんだから間違ってない。ここで階層線形モデルとかベイズ(入門編)とか色々学んでゆきました。広島から参加者が来ることもあって、しかもみんな寿司が好きで歌が上手いので、いつしか常連メンバーが増えていきました。
いいぞ、Harukara.R、どうしてそんなに大きくなってしまったんだい? 真面目にやってきたからよ、ね?(アリさんマークのひっこs)
アヒル本の読書会でもする?
そんなこんなで2016年秋。いつものように寿司を食べていた僕らは、「次のHarukara.Rでは何の勉強する?」と雑談していました。
聞くところによると、StanとRでベイズ統計モデリングという本(のちに、アヒル本という通称が生まれる)が発売になるらしい。しかも著者は、A氏が愛読しているブログの著者らしい。
じゃあもう、この本の読書会するっきゃないでしょ。というわけで当初はHarukara.Rのいち企画として読書会するつもりだったんですが、せっかくだから詳しい人や興味を持っている人もたくさん巻き込んでやろう、ということで、より大きな組織に企画をお任せすることになり、そこで生まれたのがOsaka.Stanでした。
あれよあれよという間に、色んな方にご参集いただくことができ、ついにはアヒル本の著者にまでゲストトークしていただくことになり、ここで多くの出会いが生まれ、色んなことを学びました。
なんかもう、どこに行っても同じ人いるよね
気づけば、色んな勉強会が乱立していたわけですが、どこに行っても同じ人と会うわけです。そんなこんなでいつしか、広島ベイズ塾に色んな人が集まるようになって、今に至るという。
色んな人とつながり、色んな方向から学ばせていただき、一人では知り得なかったことを知ってゆく。人と人を繋ぐ、キューピットの矢を膝に受けて、走り出していくんだ、遠い地平へーーー
(完)
【書評】PsychoPyでつくる心理学実験
本記事について
朝倉書店様と訳者の先生方より、2020年9月1日出版の、PsychoPyでつくる心理学実験をご恵投いただきました!
PsychoPyの作者自身が筆者であるため、本当に素晴らしい教科書でした。まずPsychoPyについて簡単に紹介したのち、書評を記します。
※ただ書いてから気づきましたが、書評というより要約みたいになってしまいました。まあいいか。
そもそもPsychoPyとは?
University of NottinghamのJonathan Peirce氏が作成された、「心理学、神経科学、言語学のための多彩でダイナミックな実験を作成するための、オープンソース(無料)のソフトウェアパッケージ」です(本書より直接引用)。
端的にいえば、「心理学、神経科学、言語学でよく用いられる実験課題を無料で作成できるソフト」です。Pythonをベースとしているので、名前に「Py」が付いています。
PsychoPyの作者は海外の研究者ですが、愛媛大学・十河宏行先生が日本語化や、日本語ドキュメントの執筆を行ってくださっています。
インストール方法や基本的な使用方法は、関西学院大学・小川洋和先生の解説が最も分かりやすいと思います。
これまでにも同様の目的を実現する商用ソフトはありました。しかしPsychoPyは、それらと遜色ない、いやむしろ今やそれら以上に充実した機能を備えたソフトであり、しかもタダ。作者自身が実験心理学者であるため、痒い所にちゃんと手が届く仕様。
特徴的なのは、以下の3通りの方法で実験課題を作成できること。
- Builder(プログラミングのコードを一切書く必要がないGUIで、マウス操作が主体)
- Coder(Pythonのコードを書く)
- BuilderとCoderのハイブリッド(大半はBuilderで作成し、一部の機能をCoderで実現する)
このうち、Builderの操作が本当に分かりやすく、それでいて多機能なので、学部生向けの実験実習から、プロ研究者の実験まで、幅広く用いることができます。僕も学部生向けの実験実習で長年使用していますが、上記の関西学院大学・小川洋和先生の解説に沿って実演するだけで、30分程度で基本的な機能は習得してもらえます。
僕自身でも、長年にわたりBuilderで実験課題を作成してきましたが、最近はちょっと複雑な実験課題を組むことがあるため、柔軟性の高いCoderに移行しています。PsychoPyはPythonベースのソフトと書きましたが、上述のように「パッケージ」なので、心理実験等でよく組み込まれる処理(例:刺激を200msec呈示)を行うためのPythonコードが、関数として多数用意されています。
十河先生が執筆された「心理学実験プログラミング: Python/PsychoPyによる実験作成・データ処理」を丁寧に写経すれば、Python初心者でも十分にCoderで実験課題を作成できます。
この記事も非常におすすめです。
書評
ではここから書評を記します。僕が本書の特長だと思った点は、以下の4点です。
- Builder操作に関する解説が主体
- いくつかの代表的な実験課題(例:視覚探索課題)を例に挙げながら、機能を解説
- 実験心理学の教科書(←これが良い!)
- 外部機器との連携
1つずつ説明していきます。
1. Builder操作に関する解説
上で、「Coderのほうが柔軟性が高いから、僕自身では、最近はCoderを使っている」と書きました。え、じゃあBuilderって、GUIで簡単に操作できるとはいえ、できることが限られるんじゃないの? Coderの解説書の方が需要が高いんじゃないの? と思うかもしれません。
その疑問に対する答えは、「Yes、でもそれでいいっ! それがBEST!」です(リサリサ先生の声で)。
Builderの設計思想は、GUIで任意の実験課題を作れるようにすることではありません。Builderは、GUIで「標準的な実験課題」を作れるように用意されたもの。Builderには多くのメニューが用意されていますが、意図的に機能を網羅していません。なぜなら、多すぎるメニューは利便性を低下させるし、何より初心者を混乱させるからです。幅広いユーザーが、簡便に標準的な実験課題を作れるように調整されたインタフェースが、Builderなのです。
したがって、初心者は難なく使用方法を習得できるし、プロ研究者であっても標準的な実験課題を用いる場合にはBuilderを使って何の問題もありません。むしろ素早く実験を作成できるのだから、その方がいいじゃないか。デバグも楽になるし。
あ、熱く語りましたが、これは本書に記載されていることです。僕はここを読んだとき、ハートが震えてヒートが燃えつきました(いや燃えつきたらあかん)。PsychoPyの作者自身が執筆した書籍なので、こういう作者の思いが垣間見えるのがいいですね。
ちなみに、後述もしますが、PsychoPyはオンライン実験にも対応しています。PsychoPyで作成した実験課題を専用のサーバにアップロードすることで、インターネットブラウザを介して実験を受けられるようになるということです。本記事の執筆時点(2020/8/18)では、オンライン実験用のプログラムは、原則としてCoderを使用せず、Builderで作成しなければならないと思います。そういう意味でも、Builderで課題を作成する方法の解説には意義があります。
※なお本書では、オンライン実験のための手続きは掲載されていません
2. 代表的な実験課題(例:視覚探索課題)を例に挙げながら、機能を解説
PsychoPyが見据えている研究領域(心理学、神経科学、言語学)の特徴は、「こういうデータを測定したかったら、こういう実験がよく用いられるよね」という実験パラダイムが存在することかと思います。
例えば抑制能力(特定の情報処理を我慢して、別の情報処理を行う)を測定したかったら、ストループ課題が例に挙がると思います。ストループ課題とは、「あか」「あお」のように、単語の意味とインクの色が一致する刺激(あか)と、一致しない刺激(あお)を呈示し、インクの色を回答させるという実験です(「あか」でも「あお」でも、インクの色は赤)。
あるいは、特定の情報を探し出す速さを測定したかったら、視覚探索課題が用いられると思います。例えば画面上に多くの「L」が散りばめられており、その中に「T」が1つだけ紛れ込んでいるか否かを判断するという実験です。
本書では、このような実験課題を例に挙げながら、Builderの機能を広く解説しています。汎用的な機能も学びつつ、特定の実験課題を作り上げられるので、一石二鳥ですね。
しかも、冒頭で書いたような、「作者自身が実験心理学者であるため、痒い所にちゃんと手が届く仕様」の紹介も随所に存在しています。
実験では、刺激を呈示して、それに対する被験者の反応を測定することが基本ですが、それだけすれば十分というわけではありません。ときに、測定精度を高めるための”工夫”を交えることがあります。
例えば、通常は本番の実験の前に練習を行い、被験者に実験手続きを理解していただくことが必要になります。被験者が実験手続きを理解していなかったら、本番中に測定されるデータ(例:反応の速度)は、どのような情報処理を反映しているのか分かりにくくなるからです(例:反応時間が長くても、それは刺激が処理しにくかったのか、反応方法が分からなかったのか、区別できない)。
そこで、例えば練習試行の最後の5試行ぶんだけ抽出し、反応時間や反応の正確性を確認したいというモチベーションが生まれます。そこで極端に反応が遅かったり、正答率が低かったりしなければ、被験者が実験手続きを理解したと推測することができます(もちろん、これだけで十分という意味ではありませんが)。
じゃあPsychoPyでどう実装するのか。ちゃんと実例が紹介されているんですね、これが。
このように、基本的な機能の紹介にとどまらず、「実践的テクニック」も随所に盛り込まれているのが、素晴らしい点だと思います。
3. 実験心理学の教科書
さて、僕が何より感動したのが、この点です。
ここまで、「PsychoPyを使えば簡単に実験課題が作れるよ! 実例も載ってるよ!」と、”楽ができる”ことを推してきました。しかし一般的に、「実験の準備」という手続き自体は、楽ではありません。
実験を実施する前に、丁寧に時間をかけて確認しなければならないこと、調整しなければならないことが、たくさんあります。そのために知らなければならないことが、たくさんあります。
PsychoPyは、「作る」という作業においてユーザーに楽を与えてくれます。しかし、それは諸手続きを軽視してよいことを意味しません。
例えば、現在ではPCの液晶ディスプレイを用いて実験刺激の呈示を行うことが多いと思います。一般的には、画面は60Hzで更新されることが多いでしょう(= 1秒間に60回、画面が更新される)。いわゆる、リフレッシュレートというやつです。
画面に、0.2秒だけ刺激を呈示したいとします。これは可能です。1回の更新が1/60秒なのだから、12*1/60 = 0.2であり、12回画面を更新すればよいからです。しかし同様の理屈により、画面に0.22秒だけ正確に刺激を呈示することは困難です。13.2*1/60 = 0.22なので、画面を13.2回更新すればよいわけですが、そんな半端なことはできません。
PsychoPy上で、刺激を0.22秒呈示すると設定することはできます。しかし、それは実際に刺激が0.22秒呈示されていることを意味しません。
というわけで、環境(例:使用するディスプレイ)によって実験手続きに制約が実は存在しているわけです。え...そんなの知らんかった...言うといてや...。安心してください、書いてますよ。
そもそも、液晶ディスプレイって実験で使っていいのだろうか。あれ、そういえば昔受けた実験だと、ブラウン管(CRT)モニターを使用していた気がするぞ。そもそも液晶ディスプレイとCRTモニターの違いはどうなっているのだろう? 安心してください、書いてますよ。
他にも、気を払わなければならない(かもしれない)ポイントはたくさんあります。刺激の色や輝度を厳密にコントロールしたい場合もあるでしょう。特定の情報を探す速さを測定する視覚探索課題の場合、当たり前ですが、物理的に目立つ刺激は見つけられやすくなります。こういった事情を気にする場合、キャリブレーションやガンマ補正などのキーワードが頭に浮かびます。安心してください、書いてますよ。
刺激の呈示ルールはどうすればいいだろう。とりあえずランダムな順序で呈示されるようにしておけばいいかな。でもランダム呈示って本当に万能なのかなあ。安心してください、書いてますよ。
被験者が特定の情報を認識できる/できない境界線(閾値)を知りたい。テクニックがあるのかなあ。そういえば階段法とか聞いたことがあるな...。安心してください、書いてますよ。
と、このように、実は本書は実験心理学の教科書としても非常に有用です。PsychoPyの作者自身が実験心理学者である強みが、まさにここに表れています。巻末には、三角関数の簡単な紹介も載っていて(刺激を特定の位置に呈示するためには、sinやcosの計算が必要だから)、本当に...痒い所に手が届く...。
4. 外部機器との連携
僕自身がこの用途を体験したことがないので、ここはさらっとした紹介になってしまいますが、脳活動関係(fMRI、EEG)の測定をする場合や、眼球運動の計測をする場合における、外部機器との連携が求められる発展的な機能も解説されています。これらの測定やデータの解析と相性の良い商用ソフトもありますが、フリーのPsychoPyでも実現できるのは嬉しいところだと思います。
結び
ここまでレビューしてきたように、本書は、初心者から熟練者まで、幅広いユーザーが待ち望んでいた「実験心理学の教科書」なのではないかと思います。
PsychoPyの作者自身が執筆した書籍であることによる充実度はもちろんのこと、訳者も全員、実験心理学を専門とし精力的に活躍する研究者であるため、翻訳の精度も高いと思います(まだ隅々まで精読したわけではありませんが、ざっと読んだ限りでは、非常に読みやすいと思いました)。
皆様もぜひ! Enjoy!
Windows上でzipファイルを解凍するときの、「ファイル名文字化け」への対処
本記事について
自分用メモです。
僕は論文ファイルを保存するとき、ファイル名を論文の概要にする習慣があります(この記事に感銘を受けました)。
たまった論文をフォルダごと移動させようと思って、ZIPにした後で解凍したところ、ことごとく日本語のファイル名が文字化けしました。どうやらWindowsだとこういうことが起きやすいみたいですね(理由)。
楽な解決方法が見つかったので、記録しておきます。
前提
(最近の)Windows10ではデフォルトで用意されている、Windows Subsystem for Linux(WSL)の機能を用いるなどして、Windowsマシン上でUbuntuを動かせる環境にあることを前提とします。この環境構築方法については、以下の記事が参考になります。
Ubuntu上でzipファイルの解凍
あとはもうこっちのもんです。
Ubuntu上でunarコマンドを実行すれば、解凍時に、文字コードを自動検出して良きに計らってくれると。
まずはunarをインストールして、次にunarコマンドで解凍するだけ。
sudo apt-get install unar
unar ファイル名.zip
補足
WSLとWindows間でファイルをやり取りする場合は、この方法で。
一番楽なのは、解凍対象のzipファイルを
\\wsl$\Ubuntu-(バージョン)\home\(ユーザー名)\
のディレクトリに入れた後で、上記のunarコマンドを実行することかな。
実験参加者内要因の条件間でパラメータを比較する(パラメータ間の相関を考慮した比較)
はじめに
本記事は、Stan Advent Calendar 2019 22日目の記事です。
本記事の内容はたのしいベイズモデリング 事例で拓く研究のフロンティア第15章の内容とほぼ同じですが、ちょっとだけ発展的な内容を含んでいます。
次のような手続きで実験を行ったとしましょう。PCのディスプレイ上に、「O」と「D」がランダムに呈示されます。実験参加者は、
- 「O」が出たと思ったら右のボタンを押す
- 「D」が出たと思ったら左のボタンを押す
という風に反応することを求められます。刺激が呈示されてから、実験参加者がボタンを押すまでの反応時間が記録されます。一見簡単に見えますが、これをずーーーっと続けていると、時折間違えた反応をしてしまうこともあるでしょう。また、反応が徐々に遅くなっていくと予想されます。すなわち反応の正確性や反応時間は、「情報処理が円滑に行われている程度」を反映する指標と考えられます。
モデリングの方針
このように、実験参加者に二通りの反応が許されているとき(今回は、左右いずれかのボタンを押す)、その結果として得られる反応時間のデータを、drift diffusion modelによってモデリングしてみましょう。そのようなモデリングを行う利点については、Stan Advent Calendar 2019 16日目の記事を参照してください。
本記事の目的
複数の条件を、異なる実験参加者グループが経験している場合(実験参加者「間」計画)と、同じ実験参加者が経験している場合(実験参加者「内」計画)で、モデリングの仕方が変わります。
実験参加者「内」計画の場合、同じ実験参加者がすべての条件を経験するので、条件同士で反応に相関関係が認められると予想されます。例えば反応時間を測定する実験の場合、慎重な人はすべての条件で反応が遅いかもしれないし、せっかちな人はすべての条件で反応が早いかもしれません。
本記事ではこのような実験参加者内計画における反応時間のデータを、条件間の相関を考慮したうえでモデリングする方法を紹介します。
実践
サンプルデータの作成
drift diffusion modelでは、反応時間がWiener分布に従うと考えます。そこで、Wiener分布に従う乱数を発生させましょう。brmsパッケージには、brms::rwiener()という関数が用意されています。これは内部ではRWienerというパッケージを利用しています。もし以下のコードを実行したとき「RWienerというパッケージは無い」というエラーが出た場合には、追加でRWienerパッケージをインストールしておいてください。
# load packages ---------------------------- library(tidyverse) library(brms) library(rstan) # sample data ------------------------------ trial = 1000 # 一つの条件における試行数 N_subject = 5 # 実験参加者数 tau = 0.5 # Wiener分布の非決定時間パラメータ beta = 0.5 # Wiener分布の開始点パラメータ set.seed(123) # 以下は順に、実験参加者1の条件1、条件2、実験参加者2の条件1、...、実験参加者5の条件2 dat_all = rbind(brms::rwiener(n = trial, alpha = 0.6, tau = tau, beta = beta, delta = 2), brms::rwiener(n = trial, alpha = 0.75, tau = tau, beta = beta, delta = 2.5), brms::rwiener(n = trial, alpha = 0.5, tau = tau, beta = beta, delta = 1.7), brms::rwiener(n = trial, alpha = 0.55, tau = tau, beta = beta, delta = 2.4), brms::rwiener(n = trial, alpha = 0.65, tau = tau, beta = beta, delta = 2.2), brms::rwiener(n = trial, alpha = 0.8, tau = tau, beta = beta, delta = 2.5), brms::rwiener(n = trial, alpha = 0.5, tau = tau, beta = beta, delta = 1.8), brms::rwiener(n = trial, alpha = 0.6, tau = tau, beta = beta, delta = 2.2), brms::rwiener(n = trial, alpha = 0.7, tau = tau, beta = beta, delta = 1.6), brms::rwiener(n = trial, alpha = 0.75, tau = tau, beta = beta, delta = 2.1)) %>% dplyr::mutate(ID = rep(x = 1:N_subject, each = 2 * trial), condition = rep(1:2, each = trial, time = N_subject))
Wiener分布は4つのパラメータを持ちます。このうち、非決定時間tauと、情報蓄積の開始点betaについては、今回はすべて共通としています。
反応が起こるために必要な情報蓄積の閾値alphaと、情報蓄積の速さdeltaが、実験参加者や条件によって異なるサンプルデータです。このサンプルデータには、以下のように4つの変数があります。
- q
- 反応時間(秒)
- resp
- 右のボタンを押したか(1)、左のボタンを押したか(0)
- ID
- 実験参加者のID。5人いるので、1~5。
- condition
- 「O」条件(1)、「D」条件(2)
head(dat_all) q resp ID condition 1 0.5569206 1 1 1 2 0.6966415 0 1 1 3 0.5378940 1 1 1 4 0.5503652 1 1 1 5 0.6447536 1 1 1 6 0.7017261 0 1 1 tail(dat_all) q resp ID condition 9995 0.6605005 0 5 2 9996 0.6017658 1 5 2 9997 0.6058292 1 5 2 9998 0.5311541 1 5 2 9999 0.7406046 1 5 2 10000 0.5725912 0 5 2
Stanコード
Stanにはwiener_lpdf()という分布関数が用意されているので、基本的にはここに目的変数とパラメータを書き入れるだけです。なお16日目の記事とはやや違う書き方をしています。
パラメータ名は以下のように名付けなおしています。
- 閾値
alpha→ boundary parametera - 非決定時間
tau→t_raw及びt_new- これら二つの違いは後述
- 情報蓄積の速さ
delta→ drift rate parameterd
data { int<lower=0> N; //全データ数 int<lower=0> N_ID; //被験者数 int<lower=0> N_cond; //被験者内要因の条件数 int<lower=0> ID[N]; //被験者を識別するIDが格納されたベクトル int<lower=0> cond[N]; //被験者内条件を識別するラベルが格納されたベクトル vector<lower=0>[N] RT; //反応時間ベクトル real<lower=0> minRT; //全被験者のなかでの最小RT(推定されたtがこれを超えるとエラーになるため) int Resp[N]; //反応のベクトル real<lower=0, upper = 1> beta; //starting point (0.5に固定) } parameters { vector[N_cond] a[N_ID]; //各条件のboundary parameterを計算する材料。被験者内条件数の長さで、そのベクトルが被験者数ある。 vector[N_cond] mean_mu_a; //各条件のboundary parameterが従う、多変量正規分布の平均ベクトル corr_matrix[N_cond] omega_mu_a; //各条件のboundary parameterが従う、多変量正規分布の、分散共分散行列を作るための、相関行列 vector<lower=0>[N_cond] tau_mu_a; //各条件のboundary parameterが従う、多変量正規分布の、分散共分散行列を作るための、標準偏差ベクトル real t_raw; //全被験者に共通するnon-decision time parameterを求めるための材料 vector[N_cond] d[N_ID]; //各条件のdrift rate parameterを計算する材料。被験者内条件数の長さで、そのベクトルが被験者数ある。 vector[N_cond] mean_mu_d; //各条件のdrift rate parameterが従う、多変量正規分布の平均ベクトル corr_matrix[N_cond] omega_mu_d; //各条件のdrift rate parameterが従う、多変量正規分布の、分散共分散行列を作るための、相関行列 vector<lower=0>[N_cond] tau_mu_d; //各条件のdrift rate parameterが従う、多変量正規分布の、分散共分散行列を作るための、標準偏差ベクトル } transformed parameters{ real<lower=0, upper=1> t_new; //全被験者に共通するnon-decision time parameter cov_matrix[N_cond] mu_a_covmatrix; //各条件のboundary parameterが従う、多変量正規分布の、分散共分散行列 cov_matrix[N_cond] mu_d_covmatrix; //各条件のdrift rate parameterが従う、多変量正規分布の、分散共分散行列 t_new = inv_logit(t_raw) * minRT; //ロジスティック変換で0~1に変換し、それに最小RTを掛けているため、 //Wiener分布から推定するt_newは最小RTを超えないように工夫 mu_a_covmatrix = quad_form_diag(omega_mu_a, tau_mu_a); //各条件のboundary parameterが従う、多変量正規分布の、分散共分散行列 mu_d_covmatrix = quad_form_diag(omega_mu_d, tau_mu_d); //各条件のdrift rate parameterが従う、多変量正規分布の、分散共分散行列 } model { // model ---------------------------------------------------- for(n in 1:N){ if(Resp[n] == 1){ target += wiener_lpdf(RT[n] | exp(a[ID[n], cond[n]]), t_new, beta, exp(d[ID[n], cond[n]])); //exp(a_raw), exp(v_raw)により、両parameterが多変量「対数」正規分布から生成されたことを表現 } else{ target += wiener_lpdf(RT[n] | exp(a[ID[n], cond[n]]), t_new, beta, -exp(d[ID[n], cond[n]])); } } // boundary parameter target += multi_normal_lpdf(a | mean_mu_a, mu_a_covmatrix); // drift rate parameter target += multi_normal_lpdf(d | mean_mu_d, mu_d_covmatrix); // priors ----------------------------------------------------- target += normal_lpdf(mean_mu_a | 0, 5); target += lkj_corr_lpdf(omega_mu_a | 2); target += student_t_lpdf(tau_mu_a | 3, 0, 3) - student_t_lccdf(0 | 3, 0, 3); target += normal_lpdf(t_raw | 0, 5); target += normal_lpdf(mean_mu_d | 0, 5); target += lkj_corr_lpdf(omega_mu_d | 2); target += student_t_lpdf(tau_mu_d | 3, 0, 3) - student_t_lccdf(0 | 3, 0, 3); }
ポイントは以下の点です。
wiener_lpdf()のなかで、aとdのパラメータに指数変換を施している- これらのパラメータは正の値を取ります。ということは、両パラメータに
lower=0と下限を指定するだけでもいいような気がしますが、そうすると収束が不安定になりがちでした。 aやd自体には下限を指定せず、exp()によって正の値に変換しています。
- これらのパラメータは正の値を取ります。ということは、両パラメータに
- 非決定時間パラメータ
- 反応時間の最小値を超えてしまうと推定できなくなるので、それを抑える工夫です。詳しくはコード内のコメントを見てください。
- 各条件の
aとdのパラメータが、多変量正規分布に従うと仮定している
1.と2.は推定効率を高めるための工夫ですが、3.が本記事の目的である、相関のあるパラメータを推定するために必要な部分です。
実験参加者「内」条件間でパラメータを比較したいとき、この多変量(対数)正規分布の平均ベクトルを比較することが出来ると思います。今回のモデルではmean_mu_aやmean_mu_dです。
もっとも、そういったやり方ではなく、条件間でパラメータが等しいというモデルと、パラメータが異なることを仮定するモデルを、例えばベイズファクター等を用いてモデル比較するという方法もあると思います。
推定
Rコードは以下の通りです。自己相関が高かったので、thin = 10としています。もうちょっとモデルを工夫できるのかもしれません。
# model ------------------- sm = rstan::stan_model("ddm.stan") # data -------------------- sdata = list(N = nrow(dat_all), N_ID = dat_all %>% dplyr::distinct(ID) %>% nrow(), N_cond = dat_all %>% dplyr::distinct(condition) %>% nrow(), ID = dat_all$ID, cond = dat_all$condition, RT = dat_all$q, Resp = dat_all$resp, minRT = min(dat_all$q), z = 0.5) # sampling -------------------- fit = rstan::sampling(object = sm, data = sdata, seed = 1234, iter = 8000, warmup = 3000, thin = 10)
結果
パラメータが多いので、ここでは結果の要約は載せず、パラメータリカバリできたかどうかに注目します。推定されたaやdを指数変換したものが、Wiener分布のパラメータであることに注意してください。

直線上に点が載っていたら、パラメータの真値と推定結果(パラメータの事後期待値)が一致していることを意味します。閾値aについてはうまくリカバリできたようです。
しかし情報蓄積の速さdについては、真値に近い値は得られているものの、ちょっと誤差があります。このパラメータの推定はもうちょっと工夫が要るのかもしれません。
おわりに
今回は2肢選択事態における反応時間データを、drift diffusion modelによってモデリングしました。さらにその際に、実験参加者内要因、つまり同じ実験参加者が全ての実験条件を経験したことにより、条件間でパラメータに相関が仮定される状況を取り上げました。
実験心理学ではこのような実験計画を組むことが多いので、参考になれば幸いです。
Enjoy!
R初心者の館(RとRStudioのインストール、初期設定、基本的な記法など)
本記事について
R Advent Calendar 2019 2日目の記事です。
本記事執筆のモチベーション
ゼミや講義でRを使いたいことがあります。しかし、インストールや初期設定、基本的な記法についての説明で時間を使ってしまうのはもったいないと思い、「これを事前に読んできて」と言えば済むような資料を用意したいと思いました(もちろんすでに、ネット上には有用な記事がたくさんあります)。もし同様の要望をお持ちの方がいらっしゃったら、本記事をご活用いただければ幸いです。
そういうわけで、本記事では、Rをまったく触ったことがない初心者を読者に想定しています。また、筆者の環境がWindowsであるため、同環境を事例として説明しています。
目次
RとRStudioのインストール
Rとは
公式ページの説明の通り、Rはフリーの、オープンソースのプログラミング言語で、様々な環境上で動きます(Linux系、Windows、MacOS)。統計解析や作図に強い点が特徴です。
Rのデフォルトの機能だけでも相当のことができますが、Rはパッケージ(package)を活用することで、どんどんその機能を拡張することが出来ます。
ピカチュウはでんきタイプのポケモンなので、レベルアップに伴い、基本的にはでんき系の技を中心に覚えていきます。「でんじは」とか「かみなり」とか。 しかし、わざマシンを使うと、ピカチュウがその技を使用できるようになります。例えば「ねむる」のわざマシンを使うと、自力では覚えないはずの「ねむる」を使用可能になります。
パッケージはこのように、本来備わっていない機能を、後から追加するための道具と考えてください。詳しい説明は後述します。
Rのインストール
Rにまつわるエトセトラは、CRAN(しーらん:Comprehensive R Archive Network)からダウンロードします。日本では、以下のいずれかのミラーサイトからダウンロードするとよいでしょう。
自分の環境(Linux、MaxOS、Windows)に適したリンクを選択し...

「install R for the first time」をクリックしましょう(下図の反転している部分)。最新バージョンのRをダウンロードするための画面に移動します。「Download~」をクリックするとインストーラーがダウンロードされます。


インストーラーを起動したら、「OK」と「次へ」ボタンだけを押しましょう。他の設定は一切変更しないでください。
※もし変更する場合は、インストール場所に注意してください。過去に、「パスに日本語が含まれる場所」にインストールしたり(例:C:/Users/hogehoge/Documents/分析)、同期しているクラウドストレージ(onedrive等)上にインストールしたりしたりしたため、トラブった例がありました。

これでRがインストールできました。このままRを立ち上げて操作してもいいのですが、RStudio経由でRを操作したほうが圧倒的に便利です。そこで次節ではRStudioのインストール方法を説明します。
RStudioとは
RStudioは、Rをより使いやすくするための統合開発環境(IDE)です。よく、「RStudioがあれば、Rは不要ですか?」と聞かれることがありますが、そうではありません。Rを直接操作するのではなく、RStudio経由でRを操作するわけなので、RもRStudioもそれぞれインストールする必要があります。
RStudioのインストール
ローカル環境にインストールして使用するRStudio Desktopと、サーバ上でRStudioを操作するRStudio Serverがありますが、ここではRStudio Desktopの利用方法を説明します。
RStudioの公式サイトから、最新版のRStudioインストーラーをダウンロードしてください。Installers for Supported Platformsの中から、各自の環境に合ったインストーラーを選んでください。

インストーラーを起動したら、「次へ」と「インストール」ボタンだけを押しましょう。他の設定は一切変更しないでください。
※もし変更する場合は、インストール場所に注意してください。過去に、「パスに日本語が含まれる場所」にインストールしたり(例:C:/Users/hogehoge/Documents/分析)、同期しているクラウドストレージ(onedrive等)上にインストールしたりしたりしたため、トラブった例がありました。
これでRStudioのインストールも完了です。RStudio経由でRを操作できるようになったので、以後はRStudioだけを起動させればよいです。Windowsの場合はスタートメニューから探してください。

RStudioの初期設定
次は、RStudioを本格的に使い始める前の初期設定を整えます。以後の初期設定はすべて、Tools → Global Options... の中で行います。
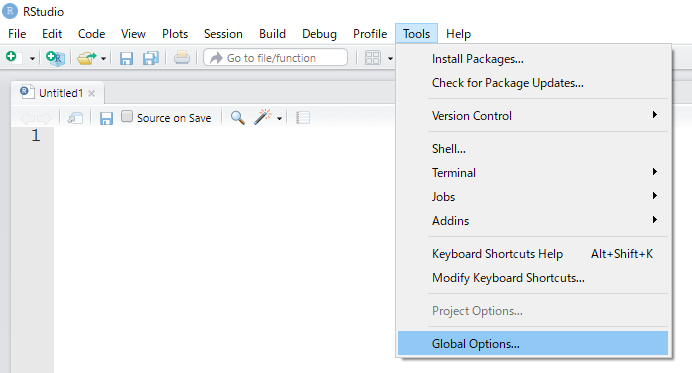
デフォルトの作業ディレクトリ(Working Directory)の設定
通常、ファイルを参照する際に、「Cドライブの中の、R_practiceというフォルダの中の、practice_1.csvという名前のファイル(C:/R_practice/practice_1.csv)」というようにファイルが置かれた場所を絶対パスで正確に指定する必要があります。いわば、郵便を送るときに、「東京都港区~~丁目の・・・さん」のように住所を書くようなものです。これは結構めんどくさいです。
しかし、作業ディレクトリを設定しておけば、その作業ディレクトリを起点とした相対パスを書けば済みます。例えば作業ディレクトリが「Cドライブの中の、R_practiceというフォルダ」であれば、いきなり「practice_1.csv」というファイル名を指定するだけで、そのファイルを特定することができます。
RStudioには「プロジェクト」という機能があり、目的ごとに作業ディレクトリを設定することができます。しかしプロジェクトが指定されていない時は、デフォルトの作業ディレクトリが適用されます。プロジェクトについては今回は説明を省略しますが、本格的にRを使うようになると必須の機能なので、その時に改めて調べてみてください。
Browse...ボタンを押して、任意のフォルダを作業ディレクトリに設定しましょう。ここではCドライブ内に「R_practice」というフォルダを新しく作り、そこをデフォルトの作業ディレクトリに指定しました。
作業ディレクトリは任意の場所で構いませんが、なるべくパスに日本語が入らないようにしたほうが安心です。次に説明するように、文字化けしたら困るので。

文字コードの設定
プログラム内に、日本語でコメントを書き入れることがあると思います。しかし日本語のようにマルチバイト文字を使用する場合、文字コードによっては文字化けが生じる恐れがあります。そこでまず文字コードを設定してしまいましょう。
左側のカラムから「Code」、上部のタブから「Saving」を選び、以下の画面を開きます。Change...ボタンを押すことで、任意の文字コードを指定できます。ここではUTF-8を選びましょう。

”見栄え”の設定
次は好みの問題ですが、好きな見栄えを選びましょう。例えばフォントサイズを変更することが出来ます。
また、RStudio上でRコードを書いた場合は、予約語やコメントに色が付いてハイライトされます。その色のパターンを、Editor themeの中から選ぶことが出来ます。

パッケージをダウンロードするCRANリポジトリの設定
先述のように、Rは次々にパッケージをインストールしていくことによって、その機能を拡張していくことが出来ます。パッケージは、世界中に存在するCRANリポジトリからインターネット経由でダウンロード&インストールすることが出来ます。その都度リポジトリを選ぶことも出来ますが、ここで設定したほうが楽です。
Change...ボタンを押すとリポジトリを変更可能です。日本国内では、以下の機関がCRANリポジトリを管理しているので、いずれかを選ぶと良いでしょう。
- 統計数理研究所(Japan (Tokyo) [https] - The Institute of Statistical Mathematics, Tokyo)
- 山形大学(Japan (Yonezawa) [https] - Yamagata University)

他にも細かい設定を行うことが可能ですが、最低限、以上の設定が終わればよいでしょう。
RStudioの機能
一度RStudioを閉じて、もう一度立ち上げなおしてください。下図の赤枠で囲ったところに、先ほど設定した作業ディレクトリが表示されていることがわかります。

さて、RStudioは上図のように、4つの領域に役割分担されています。それぞれの領域を、ペイン(Pane)と呼びます。各ペインの配置場所は変更可能ですが、ここではデフォルトの配置を前提として説明していきます。
Sourceペイン(左上)
いわゆる、スクリプトを書くところです。実はRStudioは、RもPythonもStanも...様々なプログラミング言語のコードエディタとして機能します。どのような言語であろうと、このSourceペインでスクリプトを書きます。演劇でいうところの、台本だと思ってください。
Sourceペインでは、スクリプトをただ書くだけではなく、任意のコードを即座に実行することが出来ます。実行結果は、次に説明するConsoleペインに表示されます。
コードを実行するためには、以下の操作を実行してください。MacではCtrlではなくCmdキーを使用します。
- 任意の行のコードを実行したい
- その行にカーソルを移動させて、Ctrl + Enter
- 複数行を同時に実行したい
- 複数行を選択したうえでCtrl + Enter
- あるファイル内の全ての行を実行したい
- Ctrl + Shift + Enter
Consoleペイン(左下)
上図を見るとわかるように、Sourceペインと同じコードと、その実行結果が表示されていますね。演劇でいうところの、舞台だと思ってください。
実はConsole上に直接コードを入力し、実行することも出来ます。いわば、台本にない台詞をアドリブで舞台上で披露するようなものです。しかし、分析結果の再現・再生可能性を高めるために、後で参照する可能性があるコードは全て、Sourceペインに記入したほうがよいでしょう。
Environmentペイン、Historyペイン(右上)
これらはタブでペインを切り替えます。Environmentペインには、作成したオブジェクトの一覧が表示されます。
例えば上図では、
a <- 5b <- 3
により、aというオブジェクトに5を、bというオブジェクトに3を代入しています(後述しますが、Rでは代入演算子として、矢印を示す<-を使用します)。どのオブジェクトが作成されたか、それにはどのようなデータが入っているかを、Environmentペインで確認できるというわけです。
Historyペインでは、過去に実行したコマンドの履歴が確認できます。
Filesペイン、Plotsペイン、Packagesペイン、Helpペイン、Viewerペイン(右下)
やはりこれらもタブで切り替えます。
- Filesペイン
- 作業ディレクトリ内のファイル一覧が表示されます。ここから任意のファイルを開くことも可能です。
- Plotsペイン
- 作図結果がここに表示されます。
- Packagesペイン
- 新しいパッケージを追加したい場合に、ここから選びます。また、すでにインストールされているパッケージを確認することもできます。
- Helpペイン
- 関数やパッケージ等についてのサポート情報を確認することが出来ます。
- Viewerペイン
- ちょっと特殊な出力(例えばJavaScriptベースのグラフ)が表示されます。
Rの基本的な記法・使用方法
ではいよいよ、コードを書いていきましょう。左上のアイコンを押して、新規ファイルを立ち上げます。ここではRのコードを書いていくので、一番上の「R Script」を選びましょう。このファイルの拡張子は「.R」になります。

四則演算
以下のの演算子を用いることで、四則演算が可能です。割り算の余りを返したい場合の演算子は%%です。
※注意:以後、Consoleペイン上に表示される結果を表示しています。実際のコードでは冒頭に「>」を入力する必要はありません。
※Rでは、「#」以後に書かれたものはコメントアウトされます。つまり、「#」以後に書いたものは、プログラムのコードとは認識されないため、様々なメモを残すことが可能ということです。
> 5 + 3 #足し算 [1] 8 > 5 - 3 #引き算 [1] 2 > 5 * 3 #掛け算 [1] 15 > 5 / 3 #割り算 [1] 1.666667 > 5 %% 3 #割り算の余り [1] 2
代入演算子
先述のように、Rにおける代入演算子は、矢印を表す<-です(半角の「<」の後に、半角の「-」を続けて打つ)。この代入演算子の面白いところは、->のように左右反転させても機能することです。この場合、->の左側の情報が右側のオブジェクトの中に代入されます(もっとも、この用法はあまりおすすめはしません)。
代入演算子<-は、Alt + =でショートカット入力させることもできます(+は入力する必要ありません)。
多くのプログラミング言語では、代入演算子は=ですが、Rでも使用可能です。現在のRではどちらの演算子を用いてもほぼ問題ありません。ごく稀に、<-でしか機能しない状況が存在するため、そういう意味では常に<-を使用したほうが汎用的ではあります。
> a <- 5 #aに5を代入 > a #aの中身を表示 [1] 5 > b = 3 #bに3を代入 > b #bの中身を表示 [1] 3 > 7 -> c #cに7を代入 > c #cの中身を表示 [1] 7
なお筆者はここ数年、代入演算子は=を用いています。Rを教える際に、他の言語(C、Python...)をすでに学んでいる/これから学ぶ学生が混乱しないように配慮していたのですが、いつの間にか筆者自身が=に慣れてしまいました。しかし特に困ったことはありません。
その他の演算子
Rには非常に多くの演算子があり、すべてをいきなり理解するのは難しいので、徐々に覚えていけばいいと思います。より高度な演算子は、以下のサイトを参考にしてください。
データの型・構造
とてもとてもとてもとても大事なところです。ただし、ここをちゃんと説明しようと思ったらかなり大変なので、参考となるサイトを紹介するにとどめます。ぜひ以下のリンク先を熟読してください。英語のサイトですが、非常に端的にまとまっています。ここを読むのがおすすめです。
日本語で書かれたサイトでは、以下の記事が参考になります。
ベクトルの作成
上記のサイトにも書かれている内容のうち、重要な内容を一部抜粋して説明します。まずはベクトルの作成についてです。
ここではベクトルとは、「複数の情報が並んだもの」くらいの理解で大丈夫です。例えばトランプから無作為に5枚のカードを抜き取ったところ、数字は順に「3, 1, 2, 5, 8」だったとしましょう。これもベクトルです。
Rでは関数c()によってベクトルを作成します。要素をカンマで区切って並べます。文字列を要素とする場合は、引用符("")で囲う必要がある点に注意です。
> v1 <- c(3, 1, 2, 5, 8) > v1 [1] 3 1 2 5 8 > v2 <- c("abc", "あいう", "123", "春", "-----") > v2 [1] "abc" "あいう" "123" "春" "-----"
ベクトル内の要素にアクセス
あるベクトルの中の、n番目の要素にアクセスするときは、以下のように書きます。
> #1番目(最初)の要素を表示 > v1[1] [1] 3 > # length()でベクトルの要素数を取得 > # 取得した要素数をインデックスにすることで、最後の要素を表示 > v1[length(v1)] [1] 8
ベクトル名の直後に[]を書いて、その中に数字を入力することで、対応する要素にアクセスすることが出来ます。多くのプログラミング言語では、インデックスは0から始まると思いますが(最初の要素はv1[0]、2番目の要素はv1[1])、Rでは1から始まることに注意が必要です。
matrixの作成
ベクトルが縦、あるいは横にずらっと並んだものが行列(matrix)のイメージです。例えば上で作成した2つのベクトル、v1とv2を縦方向に並べてみると...
#rbind()はベクトルを「縦方向」に連結する関数 #各ベクトルは左から右に向かってデータが並ぶ、すなわち「行(row)」になるので、頭文字r > m1 <- rbind(v1, v2) > m1 [,1] [,2] [,3] [,4] [,5] v1 "3" "1" "2" "5" "8" v2 "abc" "あいう" "123" "春" "-----"
横方向に並べてみると...
#cbind()はベクトルを「横方向」に連結する関数 #各ベクトルは上から下に向かってデータが並ぶ、すなわち「列(column)」になるので、頭文字c > m2 <- cbind(v1, v2) > m2 v1 v2 [1,] "3" "abc" [2,] "1" "あいう" [3,] "2" "123" [4,] "5" "春" [5,] "8" "-----"
いずれの場合も、個々のベクトルの行数、または列数が等しくないと連結できないことに注意してください。
> m3 <- cbind(c(1, 2, 3), c(4, 5, 6, 7, 8) ) 警告メッセージ: cbind(c(1, 2, 3), c(4, 5, 6, 7, 8)) で: number of rows of result is not a multiple of vector length (arg 1)
matrixの要素にアクセスする
matrixには行と列が存在するため、ある要素にアクセスしたい場合には、行番号と列番号をそれぞれ指定する必要があります。例えば上で作成した、m2というmatrixから「春」というデータを参照したい場合には、以下のように書きます。[行番号, 列番号]という順番であることに注意してください。
> m2[4, 2] v2 "春"
また、特定の行番号だけ、あるいは特定の列番号だけを指定すれば、該当するベクトルを参照することも出来ます。
> # 2行目だけ取得。カンマの右側が空白であることに注意 > m2[2,] v1 v2 "1" "あいう" > # 1列目だけ取得。カンマの左側が空白であることに注意 > m2[,1] [1] "3" "1" "2" "5" "8"
さらに、c()によって複数の行番号や列番号を指定することもできます。
> # 3行目と5行目だけを取得 > m2[c(3, 5),] v1 v2 [1,] "2" "123" [2,] "8" "-----"
data.frameの作成
matrixは、複数のベクトルが縦方向または横方向にずらっと並び、長方形の形になったものですが、Rでは同じ形をしているデータフレーム(data.frame)というクラスもあります。Rでは、データをdata.frameクラスにしたうえで扱うことが多いです。
data.frame()関数に複数のベクトルを与えると、data.frameを作成することが出来ます。
> df1 <- data.frame(v1, v2 = c("a", "b", "c", "d", "e"), v3 = 10:14) #c(10, 11, 12, 13, 14)に等しい > df1 v1 v2 v3 1 3 a 10 2 1 b 11 3 2 c 12 4 5 d 13 5 8 e 14
ここで、ベクトルv1はすでに作成していたものを利用しています。v2とv3は新たに作成したベクトルです。作成されたdata.frameでは、個々のベクトルに名前が与えられ(列名)、上から下にデータが並んでいます。
あたかも、cbind()でベクトルを結合した場合と同じように見えますが、クラスは異なります。
> class(m2) [1] "matrix" > class(df1) [1] "data.frame"
data.frameでもmatrixと同様に、行番号や列番号を指定することで、要素にアクセスすることが出来ます。さらにdata.frameでは列名が存在するので(もしdata.frame作成時に列名を明示しなかった場合には、適当な列名がつけられます)、列名を明示して特定の要素にアクセスすることもできます。
例えば上で作成したdf1というdata.frameで、v1列をごっそり抽出したかったら、data.frame名$列名と書きます。
> df1$v1 [1] 3 1 2 5 8
これはベクトルとして抽出されているので、さらにベクトル内の位置を指定することで、該当する要素にアクセスできます。
> df1$v1[3] [1] 2
パッケージのインストールと読み込み
インストール
PackagesペインのInstallボタンを押すと...

下図(左)のウィンドウが現れます。インストールしたいパッケージ名を入力すると、候補が現れるので、適切なパッケージを選びましょう。ここでは、データの要約や因子分析や媒介分析や...とにかく様々なことが可能になる便利なパッケージ、「psychパッケージ」をインストールしてみます。Installボタンを押すとインストールが開始されます。

あるいは以下のようにinstall.packages()関数を用いて、コードによりインストールするパッケージを指定することも出来ます。
install.packages("psych")
一度パッケージをインストールすれば、Rのバージョンが変わらない限り(つまり別のバージョンのRを再インストールしない限り)、そのPCでは同じパッケージをインストールする必要はありません。
ただし、パッケージによっては開発が続けられており、バージョンアップが行われることがあります。もし最新版のパッケージがCRAN上で配布されていれば、再び上記の手続きをとることにより、最新版のパッケージをインストールすることが出来ます。
「もし最新版のパッケージがCRAN上で配布されていれば」というところがポイントで、パッケージによっては、最新版が現時点ではCRANにまだ登録されていなかったり(いずれは登録されるようになるでしょう)、そもそもパッケージ自体がCRANに登録されていなかったりすることがあります。よくあるのは、「GitHub上ではパッケージが公開されているけれど、CRANには(まだ)登録されていない」というケースです。
その場合、
- devtoolsというパッケージをインストールして、GitHubからパッケージをインストールする機能をRに拡張させる(すでにdevtoolsパッケージをインストール済みならこの手順は省略可)
- devtoolsパッケージの関数を用いて、GitHubから、本来インストールしたいパッケージをインストールする
という手順になります。
例えばggplot2というパッケージは、CRANに登録されていますが、開発中のバージョンはGitHubから直接インストールすることが出来ます(install_github()の中は、当然パッケージごとに異なるパスを書く必要があります)。
# install.packages("devtools") devtools::install_github("tidyverse/ggplot2")
ほとんどの場合は上記のようなコードがパッケージの公式ページに記載されているはずなので、コピペすればOKです。
パッケージの読み込み
さて、パッケージはポケモンで言うところの「わざマシン」のように、後付けでRの機能を拡張するための道具だと説明しました。ただし「わざマシン」と違うところがあります。それは、パッケージは使うたびに読み込みが必要というところです。
「わざマシン」であれば、ひとたびピカチュウに「ねむる」のわざを覚えさせると、ポケモンセンターに寄ろうが、パーティーメンバーから外そうが、バトルで倒れようが、覚えたわざを使い続けることが出来ます。
しかしパッケージは、Rのセッションを立ち上げるたびに(Rを起動するたびに)、パッケージを読み込みなおす必要があります。インストールではありません。パッケージをそのセッション内で使用する場合は、読み込みが必要ということです。
例えば、psychパッケージには、ある量的変数に関する様々な要約統計量を計算してくれる、describe()という関数があります。この関数を、mtcars$mpgという変数に適用してみましょう。
> describe(mtcars$mpg) describe(mtcars$mpg) でエラー: 関数 "describe" を見つけることができませんでした
できひんのかーい。
それもそのはず。このRセッションでは、まだpsychパッケージを読み込んでいなかったからです。R側からすると、「describe()...?いえ、知らない関数ですね」ということになります。
ではパッケージを読み込みましょう。それには、library()という関数を用います。
> library(psych) > describe(mtcars$mpg) vars n mean sd median trimmed mad min max range skew kurtosis se X1 1 32 20.09 6.03 19.2 19.7 5.41 10.4 33.9 23.5 0.61 -0.37 1.07
パッケージを読み込みさえすれば、ちゃんとそのパッケージ内の関数を使えるようになりますね。
さて、ここで注意があります(次に述べることは筆者個人の意見です)。それは、Rにデフォルトで備わっている関数ではなく、パッケージ内の関数を用いる場合は、その関数がどのパッケージの関数かを常に明示すべきということです。
なぜかというと、プログラムのコードは、自分一人が見るものではなく、多くの場合は「今の自分以外」と共有するからです。「今の自分以外」には、他者も、「数年後の自分」も含まれます。
上記の例では、「describe()という関数が、psychパッケージの関数であることを知らない人」がいたら、混乱することでしょう。そこで、筆者はどのような関数であれ、パッケージ内の関数を使用する場合は、以下のように書くことを自分ルールとしています。
> library(psych) > psych::describe(mtcars$mpg) vars n mean sd median trimmed mad min max range skew kurtosis se X1 1 32 20.09 6.03 19.2 19.7 5.41 10.4 33.9 23.5 0.61 -0.37 1.07
違いがわかるでしょうか。psych::describe(mtcars$mpg)と、関数名の前にパッケージ名を明記した点だけが違います。Rではこのように、パッケージ名を明記する場合は、「パッケージ名::パッケージ内の関数」と書きます(半角のコロン2つ)。
これには別の利点もあります。Rでは、複数のパッケージが、同じ名前の関数を持っていることがあります。例えば、psychパッケージは、psych::mediate()という関数を持っていますが、別のmediationパッケージも、mediation::mediate()という関数を持っています。
関数名しか書かなかったとしたら、どちらのパッケージのmediate()関数なのか、区別がつきません。
外部ファイルの読み込み
csvファイルの読み込み
Rで解析をする際に、実験データなどがまとめられたファイルをRに読み込むことが多いでしょう。外部ファイルを読み込むための関数は、そのファイルがどのような形式でデータをまとめているかによって変わります。
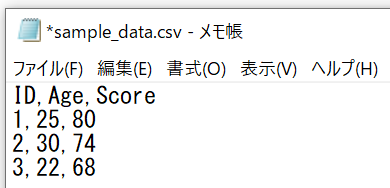
典型的には、csvファイルとしてまとめられていることが多いと思います。csvとはComma Separated Valueの略で、下図のようにカンマでデータが区切られた、テキスト形式のファイルです。
Excelでデータをまとめている場合にも、「名前を付けて保存」する際に、「ファイルの種類」をcsvに変更すれば、csvファイルとして保存されます(拡張子が.csvになっていることを確認してください)。
csvファイルを読み込む方法は、主に2つです。
一つ目は、EnvironmentペインのImport Datasetボタンから「From Text」を選択し、GUIにより読み込む方法です。「From Text」には以下の2つがあります。
- From Text (base)
- デフォルトで使用可能な
read.csv()関数によってデータを読み込む
- デフォルトで使用可能な
- From Text (readr)
readrパッケージのread_csv()関数によってデータを読み込む

どちらを使うのが良いかは話すと長くなるので、ここでは割愛します。もし数値だけが格納されたデータセットを読み込むのならば、どちらでも構わないと思います。
二つ目の方法は、直接read.csv()やreadr::read_csv()という関数をエディタやコンソール上で実行することです。実は上記のGUIを用いた場合にも、結局これらの関数が実行されることになります。
tmp <- read.csv("C:/R_practice/sample_data.csv")
注意してほしいことは、原則的に、読み込むデータセットは、長方形でなければならないということです。つまり、列によって行数が違うような凸凹したデータセットは読み込めないということです。
原則的に、と書いたのは、凸凹したデータセットでも読み込む方法があるからです。
例えば、データがこんな風にまとめられていたとしましょう。
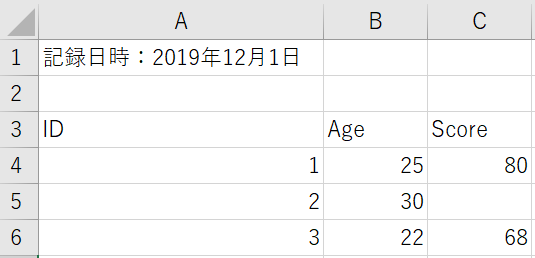
このデータセットは、以下の悩ましい問題を含んでいます。
- 1行目に、1セルだけ、分析とは関係ない情報(記録日時)が掲載されている
- 2行目が空白
Scoreの列に空白のセルがある
でも大丈夫。外部ファイルを読み込むための関数には、データを柔軟に読み込むための様々な引数が存在するのです。
tmp <- read.csv("C:/R_practice/sample_data.csv", skip = 2, na.strings = "", header = TRUE) > tmp ID Age Score 1 1 25 80 2 2 30 NA 3 3 22 68
うまく読み込むことができました。それぞれの引数を説明します。
skip = 2- 上から何行を無視するかを決めることができます。今回は、2行目までを無視しました。
na.strings = ""- データが存在しないセルを、Rでは
NAで表します。どのようなデータをNAとみなすかを指定できます。 - 今回は空白のセルを
NAとしたかったので、引用符の中に何も記述しませんでした。 - もしデータが欠測していたセルを
.で表していたなら、na.strings = "."とすればよいです。
- データが存在しないセルを、Rでは
header = TRUE- 読み込む対象の範囲のうち、一番上の行をヘッダ(列名)とみなすか、データの一部とみなすか、を指定できます。
- もし一番上の行がいきなりデータから始まっていたら、
header = FALSEとしましょう。
他にも色々と引数があります。
Excelファイルの読み込み
readxlパッケージのread_excel()という関数によって、Excelファイル(拡張子が.xlsxなど)を読み込むことも出来るようになりました。直接この関数をエディタやコンソール上で打ち込んでもいいし、やはりGUIも用意されています。
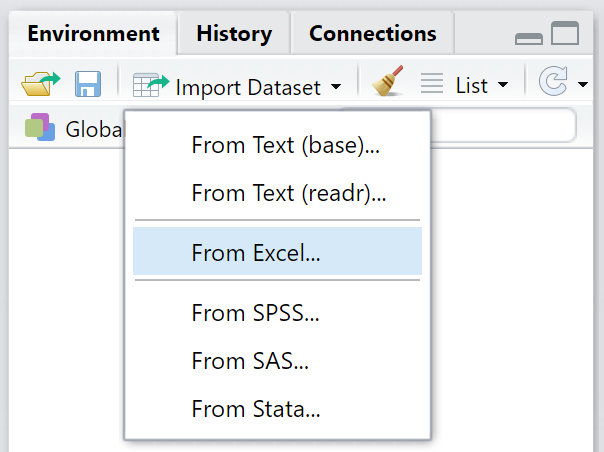
readxl::read_excel()の引数は、read.csv()やreadr::read_csv()とはちょっと異なっています。詳しくはコンソール上で?readxl::read_excelと入力して、ヘルプを確認してください。
あ、そうそう。このように、?の後ろに関数名を書くと、RStudioの右下にあるHelpペインに解説が表示されます。積極的に活用してください。

おまけ:データ構造の確認
自分でdata.frameなどを作成する場合には、どのようなデータを扱っているか(特に、数値か文字列か)について把握しやすいですが、外部からデータを読み込んだ場合は、本当に自分が想定するデータの構造になっているか、確認することが必須です。というか常に確認してください。
例えば、lme4というパッケージを読み込むと参照できるようになる、sleepstudyというデータセットがあります。これは、睡眠時間が削られることで、刺激に対する反応時間がどう変化していくかを記録したデータセットです。以下の通り3列が存在し、いずれも数値が格納されているように見えます。
> #install.packages("lme4") > library(lme4) > data(lme4::sleepstudy) > head(lme4::sleepstudy) #data.frameの冒頭6行だけ表示する Reaction Days Subject 1 249.5600 0 308 2 258.7047 1 308 3 250.8006 2 308 4 321.4398 3 308 5 356.8519 4 308 6 414.6901 5 308
ここでstr()という関数を用いて、データ構造(structure)を確認してみましょう。
> str(lme4::sleepstudy) 'data.frame': 180 obs. of 3 variables: $ Reaction: num 250 259 251 321 357 ... $ Days : num 0 1 2 3 4 5 6 7 8 9 ... $ Subject : Factor w/ 18 levels "308","309","310",..: 1 1 1 1 1 1 1 1 1 1 ...
反応時間を表すReactionや、実験経過日数を示すDaysには、numと書かれています。本記事ではデータの型や構造について説明を省略したので、何のことかわからないかもしれませんが、要は「数値」と見なされているということです。numはnumericの略です。
一方、実験参加者のID番号を示す、Subjectには、Factorと書かれています。よく見ると、"308", "309", "310",...と、各数字が引用符で囲われていますね。すなわち、見た目は数値であっても、これらは文字列のように見なされているということです。
このようなデータ構造をしっかりと確認しないと、分析時などに思わぬミスにつながります。特に外部ファイルを読み込んだ場合や、他者が作ったデータセットを使用する場合は、必ずstr()でデータ構造を確認するようにしてください。
おわりに
他にも書きたいことは山ほどありますが、キリがないのでこのあたりにします。あくまで今回の記事の目的は、「まったくRを触ったことが無い人に、事前に読んできてもらう資料」を作成することでした。これらを踏まえたうえで、他の基本的な機能や、より発展的な機能を勉強していただければ幸いです。
Enjoy!
「RとStanではじめる ベイズ統計モデリングによるデータ分析入門」書評
この記事について
著者の馬場真哉様より、2019年7月10日に講談社より発売の、「RとStanではじめる ベイズ統計モデリングによるデータ分析入門」をご恵投いただきました。ありがとうございます!!
事前に献本をいただけるということを伺っていたので、その時から「ご恵投いただきました!」とTwitterで報告するだけでなく、簡単にでも読んでみた感想を書こうと決めていました。
まだざっと読んだ段階で、コードを実際に走らせてもいないのですが(もちろん後でじっくり読みながら実行します)、感想や関連書籍との比較をしていきたいと思います。
本記事の方針
本書の「はじめに」の部分やサポートページには、以下のような方を対象読者としていると書かれています。本記事も、そのことを念頭に書いていこうと思います。
統計学の基礎やベイズの定理などの基本事項を学んでみたものの、その有効性がピンとこない、という方を対象としています。
例えば、ベイズの定理の導出では終わらない、より実用的なベイズ統計学について学んでみたいと思った方、あるいは統計モデリングに興味が出てきた方は、本書の対象読者であるといえます。理系文系は問わず、データを分析する必要性がある人はもちろん、データサイエンスが専門でないエンジニアの方でも読めるような内容を目指して執筆しました。逆に、R やStanを使ったベイズ統計モデリングにすでに明るいという方は、読者として想定していません。ベイズ統計学の数理的な側面もある程度省略しました。基本的な事項を、実装を何度も繰り返すことを通じて学ぶ構成になっています。
ベイズ統計モデリングの上級者をさらなる高みへと持っていく本ではないことに、留意してください。
また、RとStanでベイズ統計モデリングを行う入門書というと、真っ先に頭に浮かぶのが、松浦健太郎著「StanとRでベイズ統計モデリング(通称、アヒル本)」だと思います。僕自身も、恐らく人生で最も読み返した回数が多い本ではないかと思うくらい、お世話になっています。ページをめくったり書き込みしたりでボロボロになってきたので、最近2冊目を買いました。
それぞれの本の強みを紹介するため、要所要所で、アヒル本と本書を比較していこうと思います。そのため本記事の想定読者も、アヒル本を一度読んだことがある人、としています。
なお結論を先取りすると、それぞれの本で強調されている点が異なるので、両方読むといいと思います!ということになります。
第1部:【理論編】ベイズ統計モデリングの基本
本書第1部では、ベイズ統計モデリングとは何かや、それを行う上で必要となる数学的基礎知識について紹介しています。特に、ベイズ統計モデリングが基礎としている「確率」について、紙面が割かれています。
これらの基礎的なパートについては、本書とアヒル本で「力を入れている」箇所が異なるように思いました。
アヒル本
アヒル本では、確率、確率密度関数、確率質量関数、尤度などについては、ある程度背景知識を持っていることを前提として説明が進むように思います。もちろん一通り説明はあるのですが、(初心者にとっては)難しい言葉もしばしば登場します。
その代わり、モデル、確率モデル、統計モデルとは何か。そもそもなぜこれらを考える意義があるのか、といったところは紙面が割かれていて、統計モデリングに対する著者の熱い思いを感じます。「いいからMCMCだ!」ではなく、その前にすべきこと(例えばメカニズムの想像)について、慎重に指南してくれる本です。
また、アヒル本では1章まるまる使って、様々な確率分布を丁寧に紹介しているパートがあります。正規分布、ポアソン分布、二項分布などの「メジャーな」確率分布はもちろんのこと、カテゴリカル分布やディリクレ分布、多変量正規分布など、発展的なモデルを考える際にお世話になる分布についても、網羅しています。
アヒル本では、基本的な部分を順を追って丁寧に説明することを重視しているものの、基礎を身に着けた読者が自身の発想でモデリングできるよう、その先の道しるべも用意してくれているような印象を持っています。
本書
本書では、アヒル本よりもより初心者を想定しているように思います。そのため確率、確率密度関数、確率質量関数、尤度などについて、この本を読みながら知識を得ていくことが可能です。馬場さんが過去に書かれた「Pythonで学ぶあたらしい統計学の教科書」でもこのあたりは言及されています。
紹介されている確率分布も、正規分布、ベルヌーイ分布、二項分布、ポアソン分布、一様分布といった「メジャーな」ものに限定されており、より初心者にフォーカスした説明を意識されているのかなと思いました。
第2部:RとStanによるデータ分析
第2部は本書の特徴的なパートだと思います。「RとStan」でベイズ統計モデリングを行う際に必要となるRの基礎や、使用するパッケージの記法について、紹介しています。
アヒル本
アヒル本では、Rの利用方法については特に説明はなく、外部パッケージも極力使わない方針で解説されています。可視化において大活躍するggplot2パッケージは、rstanパッケージ自体が依存しているのでアヒル本でも活用されていますが、その記法は説明されていません。その代わりStanやrstanの説明に注力している感じです。
説明の順序についても、書籍間で違いがあります。アヒル本では、ページをめくるたびに、一つずつ技術を身に着けていくような説明の仕方になっている気がします。
例えばモデルの評価方法の一つである事後予測チェックは、アヒル本では簡単なモデルのパラメータ推定ができるようになった後で言及されます。
まずmodelブロックまで一通り書けるようになることを目指し、その次にgenerated quantitiesブロックの説明をする中で、事後予測チェックの方法を紹介しています(generated quantitiesブロック内で乱数を発生させる、という技術)。
本書
RからStanにデータを渡すためには、それなりにRのテクニックが必要になります。例えばlistであったり、データの型であったり。本書では最初にRの主要な機能を解説しているため、初めてRを触る人であっても問題ありません。
さらに本書は積極的に拡張パッケージを活用する方針のため、簡単ではありますがggplot2の記法を解説しています。またそれだけでなく、MCMCの結果を可視化する際にbayesplotパッケージも活用し、その使用方法も説明しています。例えば診断の類はbayesplot等の拡張パッケージに任せてしまうため、まだgenerated quantitiesブロックの話が出てきていない段階で、事後予測チェックの話が出てきます(もちろんその後でgenerated quantitiesブロックの説明も出てきます)。
※2019/7/9追記:すいません見落としていましたが、generated quantitiesブロックの中でモデルに従う乱数を生成する方法も載っていました(136ページ目)
ちなみに手前味噌になりますが、bayesplotパッケージについては、以前本ブログで記事を書いたのでご参照ください。
個人的には、bayesplotについて本書で紹介しているのは、かなり重要なポイントだと思います。診断は非常に大事な手続きだと思うのですが、診断に必要な手続きが複雑であれば、「めんどくさい」と思ってスキップしてしまうかもしれません。
実際のところ、診断の観点は様々あるのでこれ自体容易ではないのですが、少なくとも「視覚的に」診断するうえで、bayesplotはユーザの負荷を大きく下げてくれます。
第3部:一般化線形モデル
アヒル本
アヒル本では、データやパラメータをint型やreal型で宣言し、modelブロック内でforループを用いて書くシンプルな記法から始め、けっこう長い間この記法を使い続けます。ベクトルや行列を用いた書き方が登場するのは、全12章中、9章目と遅めです。
恐らくこれは、繰り返すように、「少しずつ技術を身に着けていく」ことを狙っているからではないかと思います。
本書
一方本書では、第2部でStanの基本的な記法を簡潔に紹介したのち、第3部の実践編ではいきなりベクトル・行列を活用した記法に切り替わります。説明が丁寧なので問題ないのですが、不慣れな人はここでちょっと段差が高くなった印象を受けるかもしれません。
brmsパッケージ
本書の最大の特徴と言っても過言ではないのが、brmsパッケージの活用です。brmsパッケージを用いると、ユーザ自身がStanコードを書く必要なく、シンプルな記法で線形モデルのパラメータ推定を実現することができます。
なお本ブログでも以前に記事を書いたので、ご参照ください。
このbrmsを活用したパートは、本当に現時点では稀有だと思います。僕自身が↑のブログ記事を書いたモチベーションの一つも、日本語のチュートリアル記事がほとんどないということでした。少なくとも現時点では、brmsパッケージの使用方法を事例とともにここまで丁寧に説明した和書は、他にないのではないかと思います(僕が知らないだけかもしれませんが...)。
本書第3部では、分散分析モデルやGLMの推定をbrmsパッケージに思い切って任せてしまうことで、Stanコード自体の説明はアヒル本より少なくなるけれど、そのぶん結果の可視化も含めて様々なケースを紹介しています。
特に、交互作用について独立した節を作って事例を紹介しているのは、まさに痒い所に手が届くという感じです。
第4部:一般化線形混合モデル
アヒル本
アヒル本では、階層モデルについて、「階層という発想を導入する必要性」やStanコードの書き方も含め、詳しく説明されています。Stanでは、階層モデルはよりシンプルなモデルの自然な拡張として書けるうえ、実用的機会が多いため習得することが重要だと思います。
僕自身、アヒル本を読んで初めて、「そういうことだったのか!」と目の前の霧が晴れたように理解できた部分が多かったです。
本書
一方本書では、意外なことに紙面が少ないです。それは、brmsを用いることで、階層モデル(ここでは一般化線形混合モデル)も極めて簡単に推定できてしまえるからです(lmerやlmerTestとほぼ同じ記法で書けます)。もちろんランダム切片モデルとかランダム係数モデルについての説明もあるのですが、アヒル本と対比すると説明はシンプルです。
第5部:状態空間モデル
出ました!という感じです。なにせ馬場さんといえば、時系列分析に特化した書籍、「時系列分析と状態空間モデルの基礎:RとStanで学ぶ理論と実装」の著者なのですから。
アヒル本でも状態空間モデルについて非常に分かりやすく解説されているのですが、本書では第5部がまるまる時系列データの分析に割かれています。
その代わりアヒル本では、混合分布モデルや、空間構造データを用いたモデリングなど、異なる観点で詳しくモデリング事例を紹介しています。
まとめ
冒頭にも書いた通り、アヒル本と本書は、狙いが恐らく異なっており、それぞれ強調されているポイントが違うので、両方読めば様々なケースに対応できるようになるのではないかと思いました。
あくまで僕の勝手な推測ですが、
アヒル本は、ユーザが自由なモデリングをすることを支援しているように思いました。そのために、Stanの記法について丁寧に解説し、収束しなかった場合の対処やトラブルシューティング、打ち切りや外れ値への対処、階層モデルという発想、離散パラメータを扱うテクニックなど、上級者へ至るためにいつかは通らなければならないポイントを、あらかじめ道しるべとして残していてくれているように思います。
世の中にはこういうモデルがあるんだよ、ユーザは必要なモデルを選べばいいよ、ではなく、君は君らしくモデリングする自由があるんだ(以下略)という印象です。
一方本書でも、もちろんユーザのモデリングを支援しているのですが、恐らく本書の想定読者は、アヒル本よりも初心者寄りであることから、様々なモデリングの事例をメニューとして用意してあげて、それらを導入するハードルを下げてやろう、そのためにbrmsやbayesplotなどの便利なパッケージもどんどん紹介していこう、という狙いなのかなあと思いました。
どちらか1冊読めば全方位に対応できる、ということではなく、それぞれの本が異なる強みを持っているので、これから初めて勉強する人も、すでにある程度触れている人も、両者を行き来することでさらにもう一段高みへ上れるのではないかと思います。
駆け足で読んだ段階なので、まだ見落としている部分があるかもしれません。もしかしたら今後追記するかもしれませんが、ひとまず感想まで。